JMC | 上海药物所开展卤键对蛋白质结构稳定性及其与多肽结合影响的计算研究
2024年3月19日,中国科学院上海药物研究所朱维良/徐志建团队基于数据库统计分析,并结合量子化学计算和分子动力学模拟对蛋白/多肽中的卤键进行了研究,发现在蛋白-多肽相互作用界面的卤键可以增强它们的结合亲和力,蛋白内部形成的分子内卤键有助于提高蛋白质的结构稳定性,而对于不能形成分子内卤键的蛋白质则会导致其结构稳定性降低。该研究成果以题为“Impact of Halogen Bonds on Protein–Peptide Binding and Protein Structural Stability Revealed by Computational Approaches”的文章1发表于药物化学领域著名期刊Journal of Medicinal Chemistry。该研究为卤键应用于多肽/蛋白药物的研发提供了理论性指导。
共价结合的卤素原子周围电荷分布具有各向异性,存在一个带正电的σ空穴,可以与亲核试剂相互作用形成卤键作用(XB)。卤键是分子识别和药物设计中重要的非共价相互作用。在药物设计中,已有的研究主要集中在含卤小分子化合物与靶标蛋白之间的卤键。在自然界中,一些蛋白质和肽类化合物也含有卤素原子,而卤代蛋白质和卤代肽类化合物的合成也是科研人员关注的一个领域,但目前仍然缺乏对卤代蛋白质或肽所形成的卤键的系统研究。
研究人员首先对PDB数据库中的195,093个三维结构进行了筛选,发现卤代蛋白或卤代多肽所形成的卤键主要存在于蛋白-多肽相互作用界面(inter_XBs)以及蛋白结构内部(intra_XBs)(图一a)。通过统计分析发现,侧链原子(氮和氧)比主链原子更有可能充当卤键受体(图一b),而在蛋白与小分子化合物所形成的卤键中,这种趋势并不明显2。
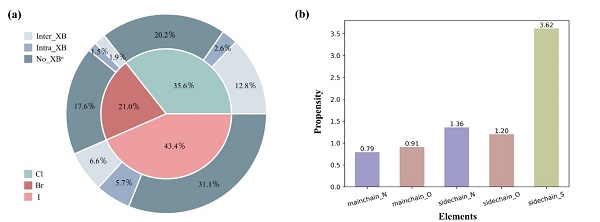
图一. 含卤蛋白/多肽的统计结果(aNo_XB表示未形成卤键)
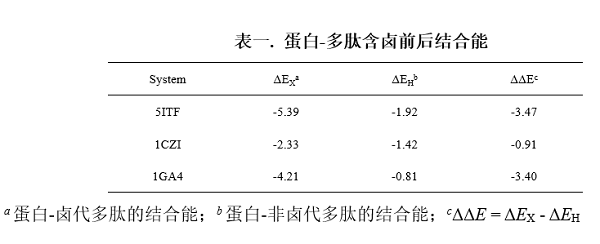
对于蛋白-多肽间的卤键,研究人员通过QM/MM结构优化、自然键轨道(NBO)理论和独立梯度模型(IGM)分析,证实蛋白-卤代多肽复合物间形成了强卤键(图二)。为了进一步研究卤键对蛋白-多肽结合的作用,研究人员对卤代前后蛋白-多肽复合物的优化结构进行了结合能计算,计算结果表明卤键的存在显著增强了蛋白-多肽间的结合作用(表一)。

图二. 蛋白-卤代多肽复合物优化前后的几何结构。初始复合物结构以灰色展示(几何参数为洋红色),优化后复合物结构以青色展示(几何参数为深蓝色)。
对于卤代蛋白质所形成的分子内卤键(intra_XBs),研究人员采用分子动力学模拟结合EP(extra point)模型对卤代蛋白的结构稳定性进行了研究,发现卤代对不同蛋白质结构稳定性的影响不同(图三)。对于结构柔性较强的蛋白,卤代对结构稳定性的影响较显著。具体地,对于卤代能形成分子内卤键的2ZXV体系,卤代后蛋白的结构稳定性增强;而对于卤代不能形成分子内卤键的5KI8体系,卤代后蛋白的结构稳定性降低。此外,对于结构刚性较强的3VN3体系,卤代对蛋白结构稳定性的影响并不明显。研究人员进一步计算了卤代前后蛋白氨基酸的局部相互作用时间(LIT)差值(ΔLIT = LITX–LITnoX),结果表明卤素的引入还会对氨基酸残基间的接触程度产生影响(图四),这应该是影响稳定性的一个重要因素。如在形成分子内卤键的2ZXV体系中,研究人员观察到卤代后氨基酸残基间的接触程度增强;而在未形成分子内卤键的5KI8体系中,卤代后残基间的接触程度减弱。
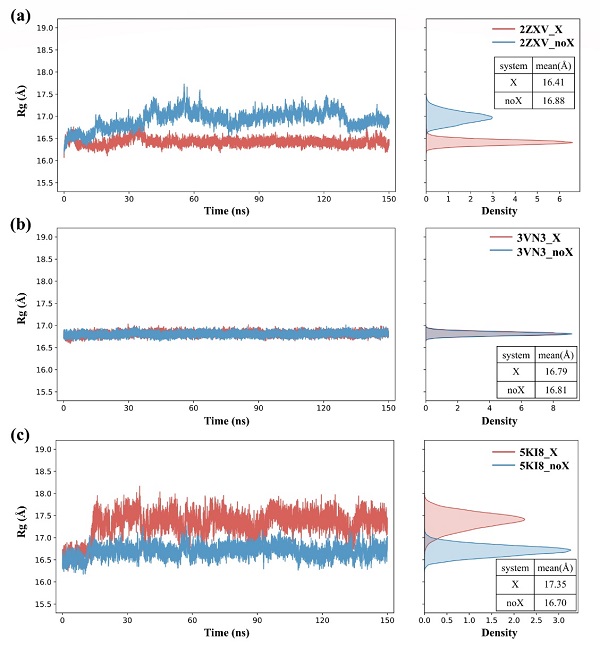
图三. 蛋白卤代前后的回旋半径分布。X表示蛋白含卤,noX表示蛋白不含卤。
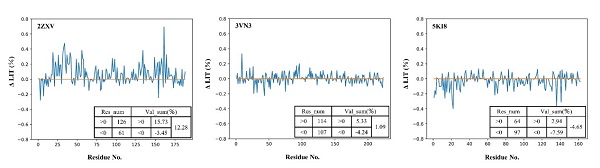
图四. 蛋白含卤前后氨基酸的ΔLIT。LIT用于衡量氨基酸与周围氨基酸的接触程度。ΔLIT = LITX–LITnoX,ΔLIT>0表示含卤蛋白中氨基酸与周围氨基酸的接触程度大于不含卤蛋白中氨基酸与周围氨基酸的接触程度。
该论文第一作者为上海药物所博士研究生李锦添,通讯作者为上海药物所徐志建研究员和朱维良研究员。该项研究工作得到了国家自然科学基金和科技部重点研发项目的资助。
徐志建/朱维良团队多年来致力于卤键领域的相关研究,首次阐明了卤键作用的本质3,揭示了PDB数据库中卤键等分子间作用力被普遍低估的现象4,首次发现核酸也可作为卤键受体5。
全文链接:https://pubs.acs.org/doi/10.1021/acs.jmedchem.3c02359
参考文献
[1] Impact of Halogen Bonds on Protein–Peptide Binding and Protein Structural Stability Revealed by Computational Approaches. J. Med. Chem. 2024, 67 (6), 4781-4792.
[2] Preferred microenvironments of halogen bonds and hydrogen bonds revealed using statistics and QM/MM calculation studies. Phys. Chem. Chem. Phys. 2023, 25 (26), 17692– 17699.
[3] Halogen bonding in differently charged complexes: basic profile, essential interaction terms and intrinsic σ-hole. Phys. Chem. Chem. Phys. 2019, 21 (27), 15106– 15119
[4] The Underestimated Halogen Bonds Forming with Protein Side Chains in Drug Discovery and Design. J. Chem. Inf. Model. 2017, 57 (1), 22−26.
[5] Halogen Bonds Exist between Noncovalent Ligands and Natural Nucleic Acids. J. Med. Chem. 2022, 65 (6), 4424−4435.