PNAS | 蛋白质-配体复合物柔性结构预测方法PackDock
近日,中国科学院上海药物研究所郑明月团队提出蛋白–配体复合物柔性结构建模新方法 PackDock。该方法将生成式AI与物理算法相结合,用于预测柔性蛋白–配体复合物构象,并在多种应用场景中展现出良好的精度与效率,同时具备较强的泛化能力。相关研究成果以 “Flexible protein–ligand docking with diffusion-based side-chain packing”为题,于2025年12月24日在Proceedings of the National Academy of Sciences of the United States of America(美国国家科学院院刊)在线发表。
理解蛋白质–配体相互作用是生命科学诸多领域的基础,从阐明细胞过程到开发疾病新疗法,都离不开对其机制的描述。作为细胞的“主力军”,蛋白质往往通过与小分子配体或其他蛋白质发生特异性结合来发挥功能。然而,蛋白质结构具有天然的动态性,配体结合也常伴随构象变化;因此,获得准确的结合构象是基于结构的药物设计的重要前提。通过在分子层面更精确地理解这些相互作用,科学家可以更深入认识生物系统如何运转,以及更有针对性地调控其功能,从而实现治疗目的。
在这项工作中,研究团队提出了柔性蛋白质建模框架 PackDock,通过整合物理建模与深度学习方法来表征蛋白质–配体相互作用。如图1所示,PackDock 同时考虑蛋白质在无配体(自由态)与配体结合态下的构象分布,从而更精准地建模柔性蛋白–配体复合物的结合构象。其核心模块 PackPocket 结合等变图神经网络与生成式建模策略,学习侧链构象空间的能量景观,并在不同状态分布中采样口袋侧链构象,以描述结合过程中可能发生的构象变化。此外,PackDock 框架可兼容多种刚性对接算法,从而进一步提升柔性对接性能。
研究团队通过多项系统性基准测试验证了PackDock 的柔性对接性能,在多种应用场景中,其整体表现优于现有分子对接算法,表明 PackDock 能够适配多来源的蛋白结构输入,并以较高精度预测蛋白–配体复合物构象。此外,在针对 ALDH1B1 的前瞻性虚拟筛选中,PackDock 成功识别出具有新型骨架且达到纳摩尔级亲和力的抑制剂,进一步证明了其在实际药物发现流程中的应用潜力。总体而言,PackDock弥补了静态结构在描绘结合过程与受体柔性方面的不足,从而推动对生物系统中蛋白–配体相互作用的机制理解。
上海药物所硕士研究生张润泽、上海药物所博士研究生江欣雨、浙江大学与上海药物所联合培养博士研究生曹端华,以及上海药物所硕士研究生王照坤为本文共同第一作者。上海药物所郑明月研究员为本文通讯作者。本研究获得国家自然科学基金、国家重点研发计划、临港实验室、中国科学院战略性先导科技专项等资助。
全文链接:https://www.pnas.org/doi/10.1073/pnas.2511925122
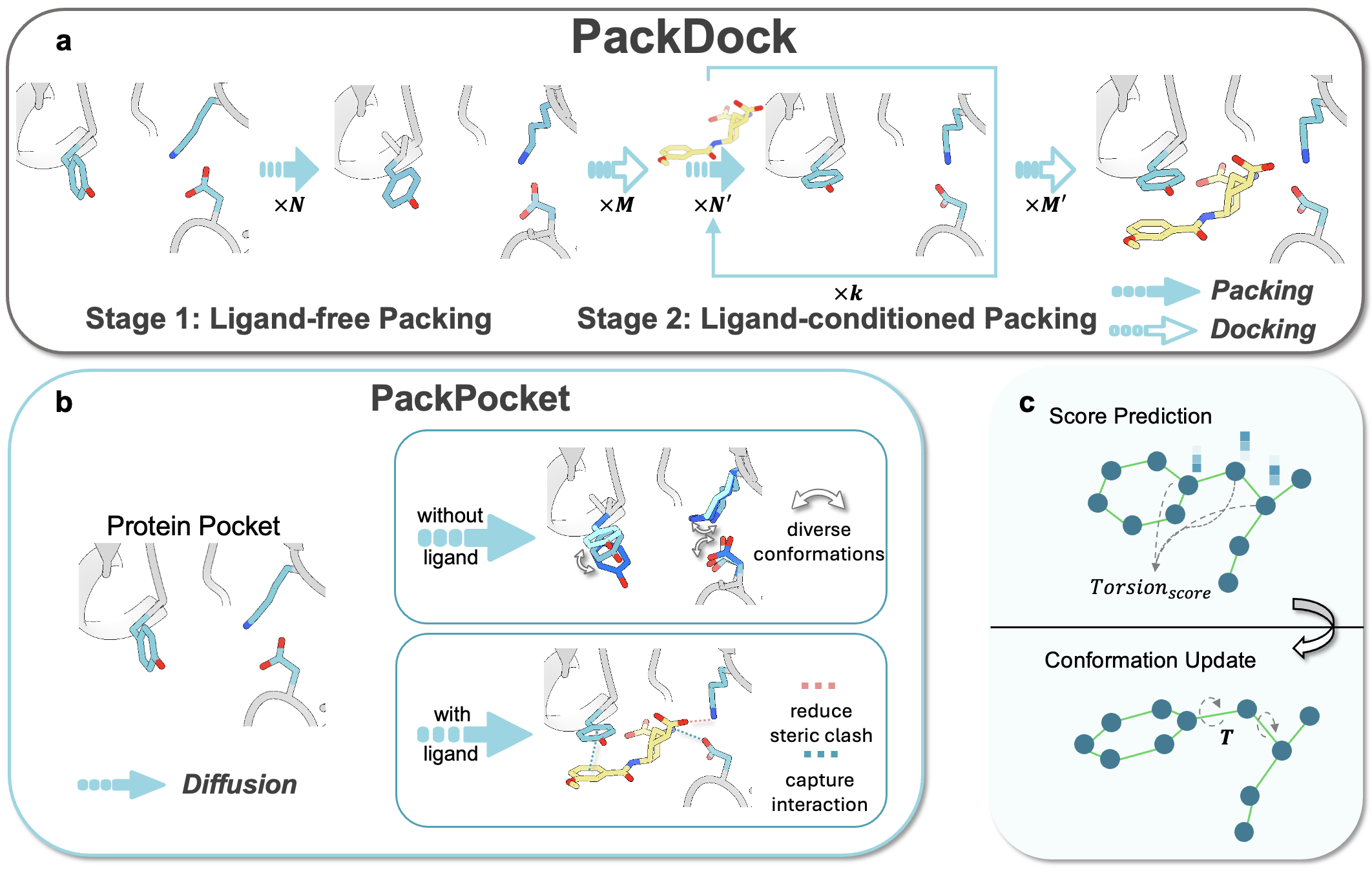
图1. PackDock 方法示意图。a:PackDock 工作流程;b:PackPocket 对侧链构象分布的建模;c:等变图神经网络预测侧链扭转角。
(供稿部门:郑明月课题组)