Brief Bioinform | 上海药物所合作构建靶向EGFR突变肺癌的用药推荐平台D3EGFR
2024年3月28日,中国科学院上海药物研究所朱维良/徐志建团队、上海市肺科医院谢冬团队和南京医科大学第一附属医院丁颖团队合作,于Briefings in Bioinformatics发表题为“D3EGFR: a webserver for deep learning-guided drug sensitivity prediction and drug response information retrieval for EGFR mutation-driven lung cancer”的研究文章。该研究通过构建表皮生长因子受体(EGFR)突变患者临床用药数据库和EGFR突变药物敏感性预测模型,有望辅助临床医生设计合理可靠的个体化治疗方案。
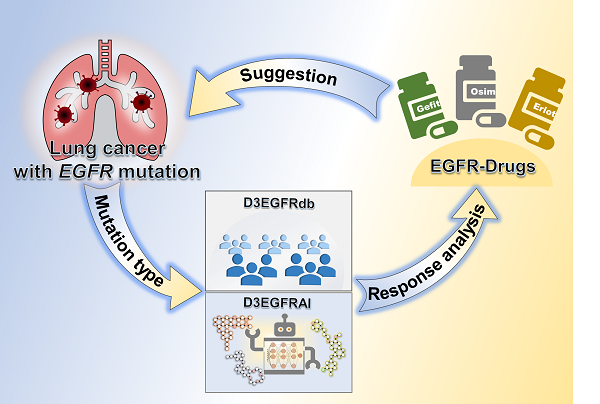
靶向EGFR突变肺癌的用药推荐平台D3EGFR
肺癌是对人类生命健康威胁最大的恶性肿瘤之一,被称为癌症的“头号杀手”。2020年发布的统计报告显示[1],肺癌在全球范围内有超过220万的新病例和超过180万的死亡人数。非小细胞肺癌是临床上最常见的肺癌类型,约占肺癌诊断总人数的85%,其主要亚型有腺癌、鳞状细胞癌以及大细胞癌。
EGFR是研究最为广泛的肺癌驱动基因之一,也是开发治疗非小细胞肺癌靶向药物的重要靶标。随着现代分子生物学技术和医疗水平的迅速发展,具有高选择性和高安全性的靶向药物成为当前肺癌精准医疗的重点研究方向。EGFR酪氨酸激酶抑制剂(EGFR-TKIs)是EGFR突变肺癌患者的标准治疗选择。但临床发现,不同突变型患者对药物的治疗效果存在差异性,且部分患者在药物治疗一段时间后会产生耐药性突变。随着基因测序方法的发展,许多临床意义不明的新型EGFR突变类型被陆续鉴定出来,给这类突变患者的个体化精准医疗带来了新的挑战。
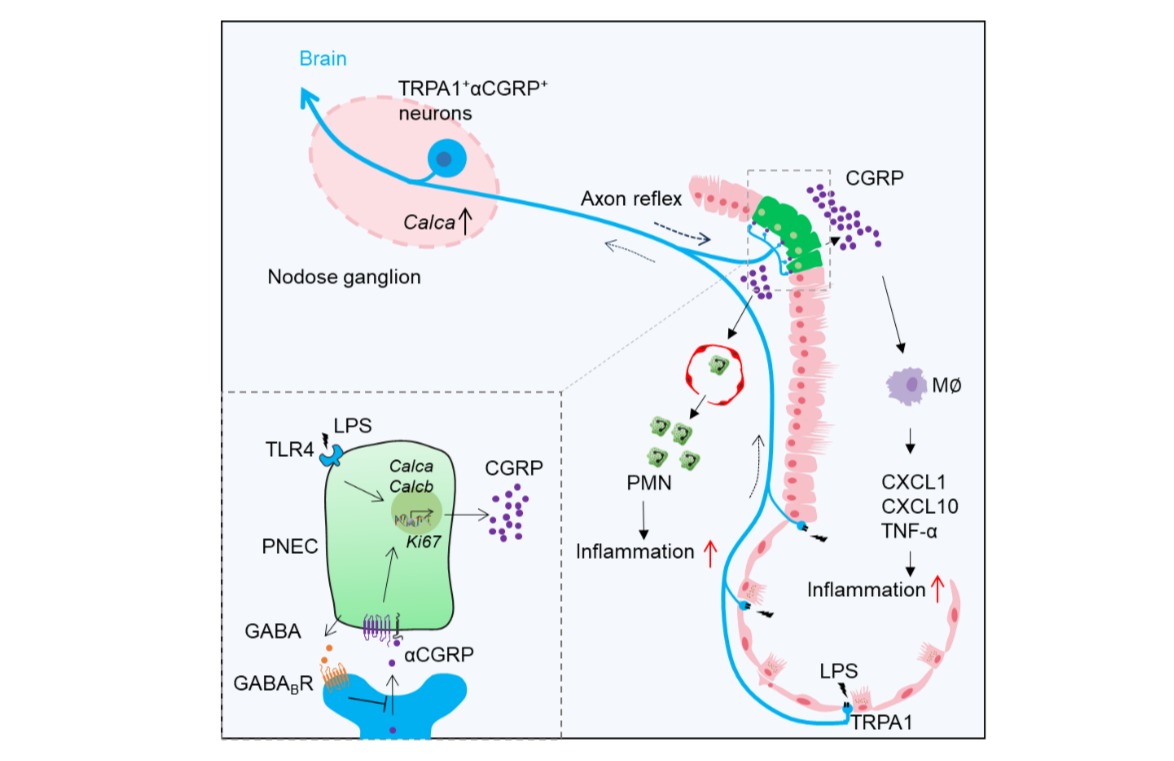
在本研究中,为了解决临床案例数据匮乏的问题,研究人员首先收集了近二十年EGFR突变肺癌患者的临床治疗相关文献,并由此构建了EGFR突变患者临床用药数据库D3EGFRdb。D3EGFRdb中收集的突变患者案例主要包含三方面信息:一是个体特征,如突变位点、性别、年龄、吸烟状况、病理学和采用的治疗药物等;二是用药结果,如药物响应、疾病进展时间(TTP)、无进展生存期(PFS)和总生存期(OS)等,药物响应类型包括完全缓解(CR)、部分缓解(PR)、疾病稳定(SD)和疾病进展(PD);三是其他信息,如临床研究类型和原始文献出处等。通过在PubMed数据库中系统检索,D3EGFRdb共收录了141篇相关文献,包含了1339例患者和257种突变类型(图1)。建立D3EGFRdb数据库的目的是:一是根据已报道的患者治疗案例为医生提供有据可循的用药决策依据;二是用于本研究中构建的计算模型的预测效果评价;三是作为未来其他研究人员开展此类相关研究的宝贵的临床数据资源。
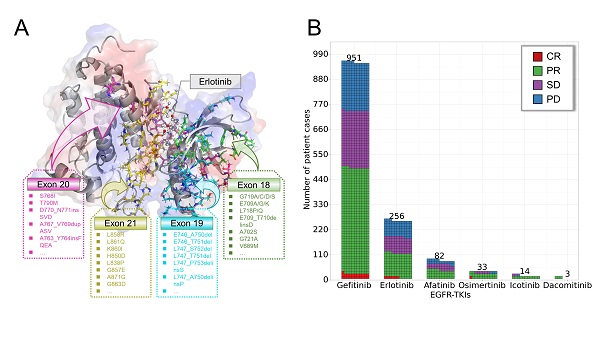
图1 D3EGFRdb中的患者突变类型和临床药物响应分布。(A)EGFR突变在蛋白三维结构上的分布;(B)每种药物的患者病例分布。
通过对临床案例进行分析(图2),女性(女性 vs. 男性:47.8% vs. 31.6%)、60-79岁(34.1%)和非吸烟者(非吸烟者 vs. 曾经或目前吸烟者:39.1%对23.8%)是EGFR突变频率较高的群体。这表明患者的个体特征与EGFR突变肺癌的发病率存在特定联系。其中,患者的主要病理是腺癌(ADC vs. 非ADC:68.1% vs. 7.9%)。在突变类型和分布位点上,点突变是最常见的突变种类(48.6%),其次是缺失型突变(16.3%),主要包括外显子21的L858R突变和外显子19的缺失突变。
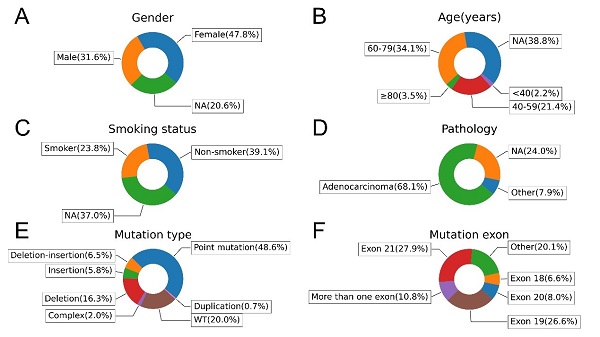
图2 D3EGFRdb中的患者个体特征。(A-F)患者的性别、年龄、吸烟史、病理学、突变类型和突变外显子分布情况。
此外,研究人员进一步考察了深度学习算法快速预测特定突变型的药物敏感性变化的可行性。深度学习算法具有神经网络架构的灵活性,在预测药物与靶标的相互作用方面取得了显著的进展。同时,深度学习模型训练过程中不依赖蛋白质三维结构,可有效避免突变体建模结构的误差影响。在该部分,研究人员采用DeepPurpose库[2]构建了80种不同编码器组合的深度学习模型,并将EGFR-TKIs药物结构和突变蛋白质的氨基酸序列及其对应的生物活性信息作为输入数据,来探索EGFR突变型与药物敏感性之间的关系。其中,有17种预测模型在生物活性数据集上的十折交叉平均相关性R大于0.8,表明利用深度学习模型预测突变蛋白质和药物的结合亲和力具有可行性。
随后,科研人员将这17种模型进一步应用于D3EGFRdb临床数据集。由于训练深度学习模型时采用的标签是生物学活性数值,而D3EGFRdb库的标签是CR、PR、SD和PD这四种药物治疗响应类型,因此需要再构建多分类逻辑回归模型,从而将深度学习模型预测的药物对不同突变型的亲和力打分与临床药物响应类别进行关联,进而处理多分类预测问题。最终发现当药物编码器为Morgan,突变蛋白编码器为CNN时,综合预测效果最佳,其在生物活性测试集上的相关性为0.86,在D3EGFRdb临床案例集上的准确率为0.81,在外部临床数据集上的准确率为0.85。基于此,将Morgan+CNN深度学习模型作为EGFR突变蛋白药物敏感性预测的最终模型,并命名为D3EGFRAI。D3EGFRAI模型构建流程如图3所示。
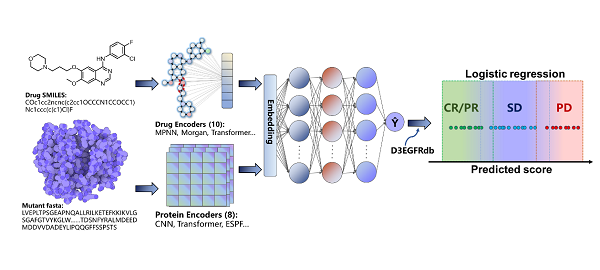
图3 不同编码器组合的深度学习框架
进一步分析临床案例,拥有同种突变型的不同患者在服用同一种药物后可能出现不同的临床药物响应。为此,D3EGFRAI模型同时输出了每种药物响应的预测概率,这将有助于更好地评估各药物的治疗效果。从中可以发现,多数突变-药物体系可能存在一到两种概率较高的药物响应(图4),这可能与患者个体差异和其他现实环境下的复杂因素有关。
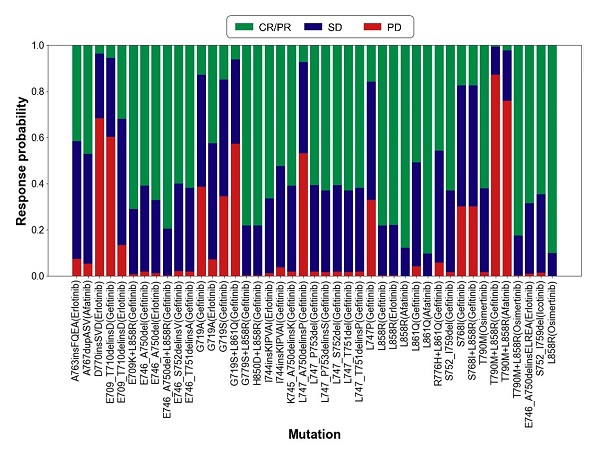
图4 不同突变体系的各药物响应预测概率
为了方便用户使用,研究人员整合D3EGFRdb数据库和D3EGFRAI模型构建了D3EGFR在线平台。该网站对所有用户免费开放,无登录要求,同时支持英文和中文(简体)语言,为用户提供EGFR突变患者的病例检索和临床药物响应预测服务。以T790M+L858R突变型为例,D3EGFRdb共收录了29例该突变型患者(图5),其中奥希替尼的CR/PR药物响应率为78.5%,优于吉非替尼(0%)、厄洛替尼(0%)和阿法替尼(14.3%),表明奥希替尼是治疗T790M+L858R突变患者的有效药物。本模块除了提供突变型与药物治疗效果的统计结果外,还提供了每位患者案例的具体临床特征和原始文献出处等信息。根据D3EGFRAI模型的预测结果,T790M+L858R突变型对第三代药物奥希替尼、艾美替尼和伏美替尼较为敏感,预测的药物响应均为CR/PR;而对第一代药物吉非替尼、厄洛替尼和埃克替尼以及第二代药物阿法替尼和达克替尼则是耐药抵抗,预测的药物响应均为PD,这一结论与D3EGFRdb的案例统计结果以及之前的报道一致。

图5 D3EGFR平台的输入和输出信息
该论文第一作者为上海药物所博士研究生石禹龙,共同第一作者为上海市肺科医院博士研究生李重武和上海药物所张鑫贲高级实验师,通讯作者为朱维良研究员、徐志建研究员、谢冬主任医师和丁颖副主任医师。该工作还得到华东师范大学张倩副研究员和南京医科大学孙鹏副教授等的大力支持。该项研究工作得到了国家自然科学基金、科技部重点研发项目等的资助。
原文链接:https://doi.org/10.1093/bib/bbae121
参考文献
[1] Sung H, Ferlay J, Siegel RL, et al.. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J Clin 2021;71:209–49.
[2] Huang K, Fu T, Glass LM, et al.. DeepPurpose: a deep learning library for drug-target interaction prediction. Bioinformatics 2021;36:5545–7.